DOI: 10.1051/gse:2000130
Genet. Sel. Evol. 32 (2000) 441-457
Cytogenetical anchoring of sheep linkage map and
syntenic groups using a sheep BAC library
Kamila Tabet-Aoula,b -
Anne Oustry-Vaimanb -
Daniel Vaimanb -
Nadhira Saïdi-Mehtarc -
Edmond-Paul Cribiub -
Frédéric Lantiera
aLaboratoire de pathologie infectieuse et immunologie,
Institut national de la recherche agronomique,
37380 Nouzilly, France
bLaboratoire de génétique biochimique et de cytogénétique,
Département de génétique animale,
Institut national de la recherche agronomique,
78352 Jouy-en-Josas Cedex, France
cLaboratoire de biologie moléculaire et génétique,
Université Oran Es-Sénia,
31000 Oran, Algérie
(Received 7 September 1999; accepted 14 March 2000)
Abstract:
In order to simultaneously integrate linkage and syntenic groups
to the ovine chromosomal map, a sheep bacterial artificial chromosome
(BAC) library was screened with previously assigned microsatellites
using a sheep-hamster hybrid panel and genetic linkage. Thirty-three
BACs were obtained, fluorescently labelled and hybridised on sheep-goat
hybrid metaphases (
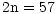
). This study allowed us, (i), to
anchor all linkage groups on sheep chromosomes, (ii), to give information
on the probable position of the centromere on the linkage map for the
centromeric chromosomes, (iii), to contradict the previous orientation
of the ovine X linkage group by the mapping of
BMS1008 on OARXq38.
Concerning our somatic cell hybrid panel, this study resulted in the
assignment of all the previously unassigned groups to ovine chromosomes
and a complete characterisation of the hybrid panel. In addition, since
hybridisations were performed on a sheep-goat hybrid, new marker/anchoring
points were added to the caprine cytogenetic map.
Keywords:
mapping / microsatellite / sheep / BAC library / FISH
Résumé:
Ancrage cytogénétique de la carte de liaison et des groupes de
synténie par utilisation d'une banque de BAC ovine.
Afin d'intégrer, la carte génétique et les groupes de synténies à la carte
chromosomique ovine, nous avons criblé, à l'aide de microsatellites, une
banque ovine de chromosomes artificiels de bactéries (BAC). Les microsatellites
utilisés font partie de la carte de liaison ovine et ont été localisés
précédemment dans notre panel d'hybrides somatiques hamster-mouton. Nous
avons isolé 33 clones BAC, marqués en fluorescence et hybridés sur métaphase
d'un hybride chèvre-mouton (
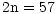
). Les résultats de cette étude
ont apporté plusieurs informations. Au niveau de la carte génétique, ces
localisations ont permis l'ancrage des groupes de liaison sur les chromosomes
ovins et de donner la position la plus probable du centromère sur les groupes
de liaison correspondant aux chromosomes métacentriques. De plus, pour le
chromosome X, la localisation du microsatellite
BMS1008 a permis de
corriger l'orientation du groupe de liaison. Au niveau de notre panel d'hybrides
somatiques hamster-mouton, ces résultats ont permis d'assigner cytogénétiquement
tous les groupes de synténies à un chromosome ovin, aboutissant à une
caractérisation approfondie du panel. Enfin, en utilisant des métaphases
d'hybride chèvre-mouton pour les hybridations
in situ, nous avons
pu ajouter de nouveaux marqueurs et points d'ancrages sur la carte chromosomique
caprine.
Mots clé :
cartographie / microsatellite / mouton / banque BAC / FISH
Correspondence and reprints: Edmond-Paul Cribiu
e-mail: cribiu@biotec.jouy.inra.fr
Copyright INRA, EDP Sciences


